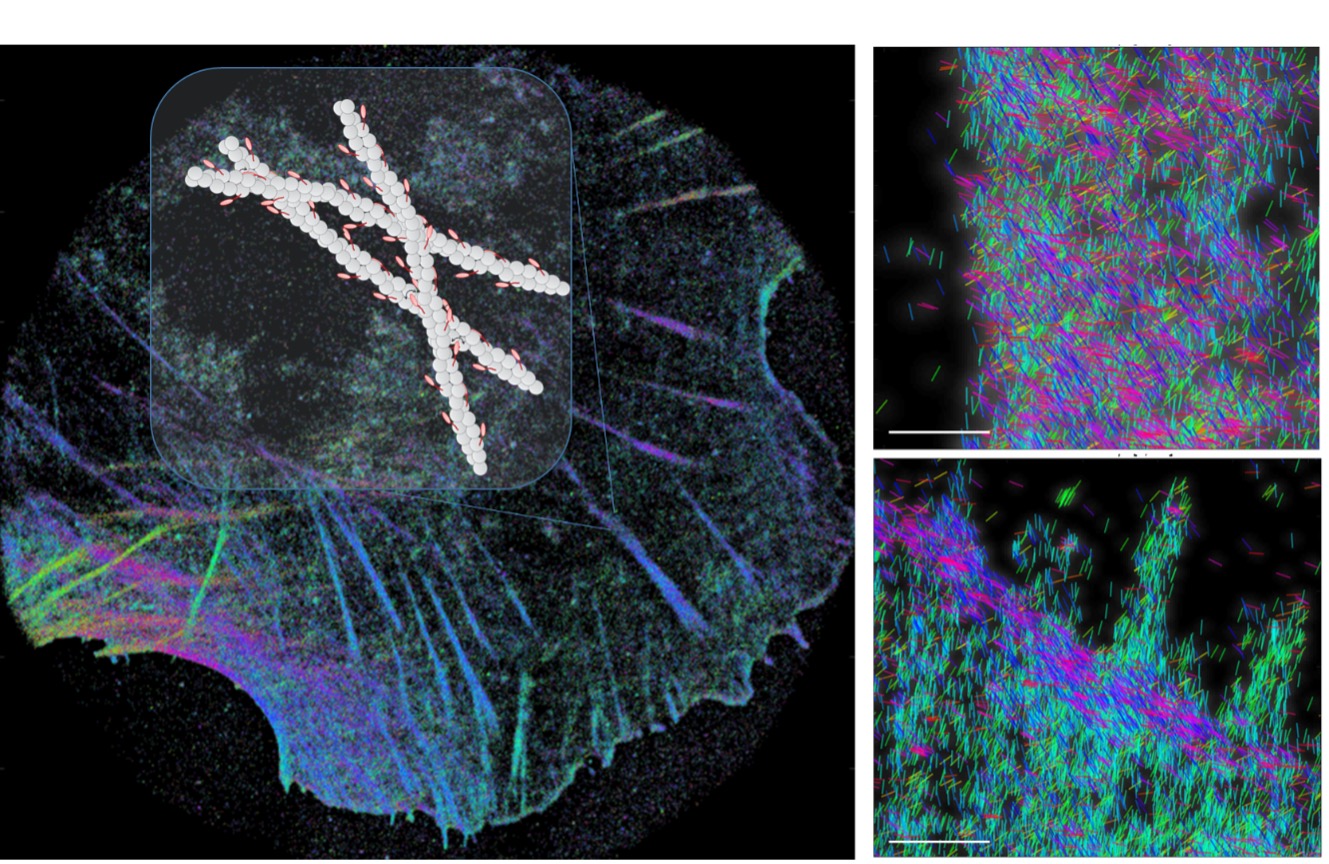
Une nouvelle approche d’imagerie polarisée à base de localisation de molécules isolées fluorescente permet de révéler l’organisation de l’actine dans les cellules aux échelles nanométriques
Imager la manière dont sont orientées les protéines en 3D dans une cellule, tout en les positionnant à des dizaines de nanomètre de précision, est un défi pour l’imagerie optique. C’est pourtant ce qui permettrait de comprendre les processus qui relient les conformations et organisation des protéines, aux fonctions qu’elles exercent dans les cellules. Alors que quelques méthodes émergent aujourd’hui pour mesurer à la fois la position et l’orientation de molécules uniques fluorescentes dans des échantillons in vitro, ces approches sont encore limitées pour l’utilisation dans des environnements complexes comme les cellules, où les conditions de signal à bruit et de fond de fluorescence sont délicates. Une des raisons principales est que ces approches se basent sur un codage de l’information de l’orientation moléculaire dans la forme de l’image de chaque molécule, forme qui peut dépendre fortement des aberrations optiques produites dans le microscope et par l’échantillon. Les chercheurs de l’équipe MOSAIC de l’Institut Fresnel sont parvenus à mettre en place une stratégie basée uniquement sur une mesure de rapports d’intensités d’une même molécule, projetée sur quatre directions de polarisation. En concevant un système de détection optique de telle manière que ces signaux sont peu dépendants des paramètres qui pourraient biaiser les données, les chercheurs ont montré la possibilité de remonter à l’information de l’orientation de molécules marquant l’actine, un filament du cytosquelette des cellules. Ces filaments, par leur densité, sont parfois impossible à isoler sur une image, même super–résolue. Grâce à cette détection polarisée, il a été possible de mettre en évidence la manière dont les filaments s’organisent avec des orientations soit fortement parallèles, comme dans les fibres de stress, soit dans des distributions beaucoup plus complexes mais spécifiques comme dans le front de migration des cellules où l’actine est agencée de manière singulière. Ces images, bien qu’étant mesurées dans des conditions optiques donc compatible avec le vivant, se rapprochent d’une imagerie structurale, révélant la manière dont les protéines s’organisent à des échelles nanométriques.

Cette nouvelle approche, appelée 4polar-STORM (pour Stochastic Optical Reconstruction Microscopy polarisée sur 4 voies), est basée sur un lien étroit entre l’orientation du marqueur fluorescent et celle de la protéine qu’ils rapportent. En développant une ingénierie des liaisons entre les marqueurs fluorescents et ces protéines, il sera possible dans le futur de suivre, dans des échantillons vivants, la manière dont ces protéines modifient leur orientation au cours du temps. L’approche de microscopie super-résolue polarisée vient donc compléter l’imagerie super-résolue de fluorescence pour aborder de nouvelles questions en biologie sous un angle différent.
Référence : V C. Rimoli, C. Valades Cruz, V. Curcio, M. Mavrakis, S. Brasselet. 4polar-STORM polarized super-resolution imaging of actin filament organization in cells. Nat. Communications 13, 301 (2022)
– https://doi.org/10.1038/s41467-022-27966-w
Financements : France BioImaging National Infrastructure (ANR-10-INBS-04), CENTURI convergence institute (ANR-16-CONV-0001), ANR SEPTIMORF (ANR-17-CE13-0014), ANR 3DPolariSR (ANR-20-CE42-0003). A*MIDEX (ANR-11-IDEX-0001), European Union’s Horizon 2020 research and innovation programme under the Marie Sklodowska-Curie (grant agreement No 713750), INRIA (projet NAVISCOPE-IPL).
Contact Chercheurs : Sophie Brasselet and Manos Mavrakis – Equipe MOSAIC, Institut Fresnel, Marseille

